納品物:
4 μg の凍結乾燥プラスミド(1 μg、低コピープラスミド用)*
電子ベクターマップ
*プラスミドコピーをご確認ください
品質管理:
お客様のカスタムインサートを含むシーケンスクロマトグラム
品質保証証明書

ホーム » CRISPRサービス » CRISPR プラスミド
GenScriptは、CRISPR技術のライセンスを、Broad Institute of MIT and Harvardより取得しています。当社の提供品には、CRISPRのパイオニアであるFeng Zhang研究室によって開発された、最新のCRISPRプラスミドおよびデータベースがあります。Broad Institute検証済のプラスミドはCRISPR/Cas9発現用の十分に試験されたプラットフォームで、またRNAベースのプラットフォームにおける不安定性の問題を回避します。
GenScript CRISPRプラスミドコレクション
GenScriptは、Broad Instituteに事前検証されたガイドRNA(gRNA)配列を含む、20,000点以上のlentiCRISPRv2プラスミドを維持しています。プラスミドは遺伝子名、シンボルあるいはIDで、当社gRNAデータベースから検索可能です。
| Product | Vector | Selection | |
|---|---|---|---|
| GenCRISPR™ Plasmid Collection | Lentiviral | Amp, Puro | Broad gRNA Database |
Enhanced CRISPR/SpCas9New!
SpCas9(K848A/K1003A/R1060A)とも呼ばれる、特異性がEnhanced CRISPR/SpCas9 (eSpCas9)は、Broad InstituteのFeng Zhang研究室の研究者らにより、ターゲットへの特異性が向上する構造的改変がされています(Slaymaker et al. 2016)。eSpCas9は、しっかりとしたオンターゲットゲノム編集効率を維持しながらも、オフターゲット効果を10倍以上低減できます。
詳細情報 »
Cas9 genome editing is dependent on the separation of DNA double strands. Mismatches between sgRNA and untargeted DNA sequences can cause unspecific binding and cleavage. To improve genome editing specificity, SpCas9 with mutations K848A, K1003A, and R1060A was developed. Neutralization of these positively charged residues within the non-target strand groove of SpCas9 weakened non-target binding and encouraged on-target binding which requires more stringent Watson-Crick base pairing.
| Product | Vector | Selection | |
|---|---|---|---|
| eSpCas9 Plasmids | Plasmid Lentiviral |
Puro or GFP | お問合せ |
SpCas9プラスミド
Cas9エンドヌクレアーゼは、遺伝子編集における研究スタンダードです。シングルガイドRNA(sgRNA)配列と組み合わせた際、これらの酵素はゲノム中に部位特異的な二本鎖切断(DSBs)を生じさせます。
詳細情報 »
SpCas9 also contains on-target affinity for NGA sequences.
| Product | Vector | Selection | |
|---|---|---|---|
| SpCas9 Plasmids | Plasmid Lentiviral AAV |
Amp Amp, Puro Amp, Neo Amp, GFP |
お問合せ |
SpCas9ニッカーゼプラスミド
SpCas9ニッカーゼ(Cas9n D10A)は、DSBsとは対照的に、エンドヌクレアーゼにより一本鎖のニックを生じることができる変異を含んでいます。二つの反対側に向いたgRNA配列をSpCas9ニッカーゼ配列と組み合わせることは、不要なインデルの形成を防ぐ、効率的な遺伝子編集の方法です。
| Product | Vector | Selection | |
|---|---|---|---|
| SpCas9 Nickase Plasmids | Plasmid Lentiviral |
Amp Amp, Puro Amp, GFP |
お問合せ |
SaCas9プラスミド
黄色ブドウ球菌Cas9オルソログ(SaCas9)は、アデノ随伴ウイルス(AAV)のアプリケーションにふさわしいエンドヌクレアーゼです。SaCas9はSpCas9よりも約1 kb短く、またAAVのパッケージングにおける制限などに、さらなる柔軟性を提供します。AAVベクターのより低い免疫原性により、SaCas9はin vivoでの編集アプリケーションや治療に最適です。
詳細情報 »
The optimal on-target SaCas9 PAM sequence is NNGRRT.
SaCas9 also contains significant on-target affinity for NNGRRN.
| Product | Vector | Selection | |
|---|---|---|---|
| SaCas9 Plasmids | AVV | Amp | お問合せ |
転写活性化(SAM)プラスミド
CRISPR/Cas9相乗的活性化メディエーター(SAM)システムは、ダウンストリームターゲットの転写活性化を可能にするよう改変されています。SAMシステムは、触媒的に活性のないCas9(dCas9)複合体上に組み立てられた3つの異なる活性化因子:VP64、P65、およびHSF1を利用して、転写を促進します。
詳細情報 »
The SAM complex is comprised of three components: a gRNA incorporating two MS2 RNA aptamers, a catalytically inactive dCas9-VP64 fusion protein, and a MS2-P65-HSF1 activator fusion protein.
The SAM system is capable of activation of both coding and non-coding genetic elements.
To search for Broad Institute pre-validated SAM gRNA sequences, visit our guide RNA Database.
| Product | Vector | Selection | |
|---|---|---|---|
| SAM gRNA Plasmids | Plasmid Lentiviral |
Amp Amp, Zeo |
お問合せ |
| SAM dCas9-VP64 Plasmids | Lentiviral | Amp, Blast Amp, GFP |
お問合せ |
| SAM MS2-P65-HSF1 Plasmids | Lentiviral | Amp, GFP Amp, Hygro |
お問合せ |
Broad Instituteプラスミドコレクション
Broad Instituteプラスミドは、HarvardとMITのBroad Instituteによって作られました。これらのプラスミドは、カスタマイズされたsgRNA配列の代わりに17 bp-1.8 kbの発現可能なリンカーを含んでおり、お客様の研究室で改変することができます
CRISPRベクター仕様
| サービス | ベクター | Cas9/gRNA発現 | 納品方法 | ベクターの種類 | 選択マーカー |
|---|---|---|---|---|---|
| eSpCas9 Plasmids | eSpCas9-2A-GFP (PX458) New! | eSpCas9 & gRNA | Plasmid | All-in-one Vector | AmpR EGFP |
| eSpCas9 Plasmids | eSpCas9-2A-Puro (PX459) V2.0 New! | eSpCas9 & gRNA | Plasmid | All-in-one Vector | AmpR PuroR |
| eSpCas9 Plasmids | eSpCas9-LentiCRISPR v2 New! | eSpCas9 & gRNA | Lentiviral | All-in-one Vector | AmpR PuroR |
| SpCas9 Plasmids | pSpCas9 BB-2A-GFP PX458 | SpCas9 & gRNA | Plasmid | All-in-one Vector | AmpR EGFP |
| SpCas9 Plasmids | pSpCas9 BB-2A-Puro (PX459) v2.0 | SpCas9 & gRNA | Plasmid | All-in-one Vector | AmpR PuroR |
| SpCas9 Plasmids | pLentiCRISPR v2 | SpCas9 & gRNA | Lentiviral | All-in-one Vector | AmpR PuroR |
| SpCas9 Plasmids SpCas9 Nickase Plasmids |
pLentiGuide-Puro | gRNA Only | Lentiviral | Dual Vector | AmpR PuroR |
| SpCas9 Plasmids | pLentiCas9-Blast | SpCas9 Only | Lentiviral | Dual Vector | AmpR BsdR BleoR |
| SpCas9 Plasmids | pLentiCas9-EGFP | SpCas9 Only | Lentiviral | Dual Vector | AmpR EGFP |
| SpCas9 Plasmids | pGS-gRNA | gRNA Only | Plasmid | Dual Vector | AmpR |
| SpCas9 Plasmids | pGS-gRNA-Neo | gRNA Only | Plasmid | Dual Vector | AmpR NeoR |
| SpCas9 Plasmids | pSpCas9 PX165 | SpCas9 Only | Plasmid | Dual Vector | AmpR |
| SpCas9 Plasmids | pAAV_SpGuide acceptor (PX552) | gRNA Only | AAV | Dual Vector | AmpR EGFP |
| SpCas9 Plasmids | pAAV-SpCas9 PX551 | SpCas9 Only | AAV | Dual Vector | AmpR |
| SpCas9 Nickase Plasmids | pSpCas9n BB PX460 | SpCas9 Nickase & gRNA | Plasmid | All-in-one Vector | AmpR |
| SpCas9 Nickase Plasmids | pSpCas9n BB-2A-GFP PX461 | SpCas9 Nickase & gRNA | Plasmid | All-in-one Vector | AmpR EGFP |
| SpCas9 Nickase Plasmids | pSpCas9n BB-2A-Puro (PX462) V2.0 | SpCas9 Nickase & gRNA | Plasmid | All-in-one Vector | AmpR PuroR |
| SpCas9 Nickase Plasmids | pLentiCas9n-Blast | SpCas9 Nickase Only | Lentiviral | Dual Vector | AmpR BsdR BleoR |
| SaCas9 Plasmids | pX601_AAV | SaCas9 & gRNA | AAV | All-in-one Vector | AmpR |
| Transcription Activation (SAM) | pSgRNA(MS2) | gRNA Only | Plasmid | SAM Multi Vector | AmpR |
| Transcription Activation (SAM) | pLenti_sgRNA(MS2)_zeo | gRNA Only | Lentiviral | SAM Multi Vector | AmpR ZeoR BeloR |
| Transcription Activation (SAM) | pLenti_dCas9-VP64_Blast | Cas9 Activator | Lentiviral | SAM Multi Vector | AmpR BlastR BleoR |
| Transcription Activation (SAM) | pLenti_dCas9-VP64_GFP | Cas9 Activator | Lentiviral | SAM Multi Vector | AmpR EGFP BleoR |
| Transcription Activation (SAM) | pLenti_MS2-P65-HSF1_Hygro | Activator Adapter | Lentiviral | Multi Vector | AmpR HygroR BleoR |
| Transcription Activation (SAM) | pLenti_MS2-P65-HSF1_GFP | Activator Adapter | Lentiviral | Multi Vector | AmpR EGFP BleoR |
SpCas9ベクター
化膿性連鎖球菌のII型CRISPRシステムに由来するCas9エンドヌクレアーゼ(SpCas9)は、哺乳類ゲノム編集向けに開発された最初のCas9酵素です。ガイドRNA(gRNA)配列と組み合わせた際、これらの酵素はゲノム中に部位特異的二本鎖切断(DSBs)を生じさせます。CRISPR/Cas9システムは、その使いやすさ、特異性、および高効率の面から、ゲノム編集を加速しました。
GenScriptはBroad Institute検証済のWT SpCas9コンストラクトを、哺乳類細胞の遺伝子編集用に提供しています。
コンストラクトには、オールインワンまたはデュアルベクターシステムがあり、非ウイルス、レンチウイルス、もしくはアデノ随伴ウイルス(AAV)トランスフェクションに使用できます。レンチベクターは第2世代および第3世代のレンチウイルスパッケージングプラスミドに対応しています。
SpCas9ニッカーゼベクター
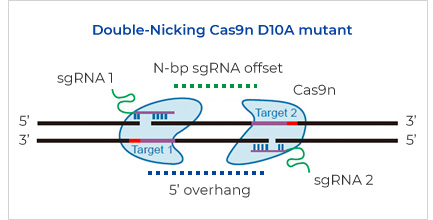
他の一般的な遺伝子編集戦略と比べ、CRISPR/Cas9技術はより特異的ですが、オフターゲット効果の懸念は、まだ現実として残っています。特異性改善のために、Cas9のエンドヌクレアーゼ活性が改変されています。WT Cas9は二つの触媒ドメインのRuvCとHNHを持ち、これらドメイン内の触媒残基への変異(特にRuvCのD10AとHNHのH840A)は、Cas9が二本鎖を切断するのに対して、一本鎖のニックを生じさせます(Ran et al, 2013)。ニッカーゼ活性を持つこのCas9酵素、またはCas9nは、ガイドRNA(gRNA)によりターゲットのゲノムDNAの反対側にガイドされます。細胞は、NHEJよりもHDRで優先的に、これらのSSBsを修復します。HDRメカニズムを通して、オフターゲットDSBsからの不要なインデル変異の頻度が最小限になります。GenScriptはBroad検証済ニッカーゼベクターを、哺乳類細胞種の遺伝子編集向けに提供しています。
SaCas9ベクター
黄色ブドウ球菌由来のCas9オルソログ、またはSaCas9は、SpCas9と同様の効率がありますが、SaCas9は約1 kb短くなっています。SaCas9の主な優位点は、アデノ随伴ウイルス(AAV)のパッケージングです。AAVの積荷容量は約4.5 kbのため、SpCas9をこのベクターにパッケージングするのは困難なことがあります(Ran et al, 2015)。比較的小さいサイズのSaCas9は、AAVベクターによるCRISPR遺伝子編集を可能にしています。 これらコンストラクトの低い免疫原性から、SaCas9は治療などのin vivo編集アプリケーションにより適しています。
転写活性化(SAM)ベクター
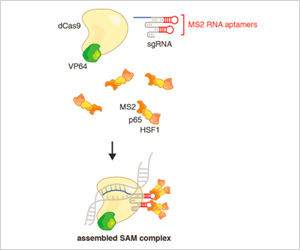
CRISPR/Cas9相乗的活性化メディエーター(SAM)は、一度に一つの遺伝子、または同じ細胞内で最大10の内在性遺伝子を同時に、安定した転写の活性化を可能にするよう改変した、タンパク質複合体です。SAMは、ガイドRNAにより内在性ゲノム中の特定の座位をターゲットとするCas9ヌクレアーゼの特異性と、リプログラミングの容易さを上手に利用しています。
Broad Institute*からのライセンスにより、GenScriptは、ヒトゲノム中のどの翻訳領域でもターゲットする検証済SAM gRNA配列および他種向けのSAM gRNAの補完的なデザインを提供しています。
SAMガイドRNA配列はカスタム合成され、効率的なレンチウイルスベクターにクローニングされます。また3つの部位でSAM複合体を形成するCas9-VP64 やMS2-P65-HSF1コンポーネントも付随します。
3つのコンポーネントで構成されるSAM複合体
1.核酸分解的に不活性なCas9-VP64融合: dCas9は、内在性ゲノムに鎖の切断が導入されないように使用されます。VP64は転写活性化ドメインで、p65やHSF1と相乗して働き、転写を増強します。
2.テトラループとステムループの2つにMS2 RNAアプタマーを組み込んだsgRNA: 理想的な転写活性化に、SAM複合体をターゲットするため、sgRNAは転写開始部位の最初の200 bp上流をターゲットするようデザインする必要があります。sgRNAは通常Cas9と結合しますが、MS2 RNAアプタマーはSAM複合体の三番目のメンバーがCas9-sgRNA複合体と結合できるようにする必要があります。
3.MS2-P65-HSF1活性化ヘルパータンパク質: このタンパク質には二つの転写活性化ドメインであるP65とHSF1が含まれており、VP64との相乗作用により、下流翻訳領域をしっかりと転写活性化します。MS2ドメインは、そのヘルパータンパク質がsgRNA-dCas9複合体と結合することを可能にします。
CRISPR Handbooks and Protocols

CRISPRプラスミド 質問と回答
納品物:
4 μg の凍結乾燥プラスミド(1 μg、低コピープラスミド用)*
電子ベクターマップ
*プラスミドコピーをご確認ください
品質管理:
お客様のカスタムインサートを含むシーケンスクロマトグラム
品質保証証明書
ノックアウトと実験の正確性を確実にするため、最低3つのgRNA配列が推奨されています。独立して得られたノックアウト変異体は、隠れたオフターゲット効果から守るための冗長性を提供します。
お客様のgRNA配列のデザインには、4つの段階が含まれます:
ターゲット遺伝子座位を決定する.
Cas9のターゲティングに適した配列を見つけ出す.
オフターゲット結合の可能性を確認する.
お客様がご希望の結合領域内にあるgRNA配列を選択する.
GenScriptのgRNAデータベースとオンラインデザインツールは、オフターゲットスコアと染色体の位置を提供することにより、お客様がgRNA配列を選択する際の、多くの推量を無くします.
オールインワンベクターシステムは、二つの利点があります:
C細胞のトランスフェクションが一度のみで済む.
gRNA/Cas9発現が、理想的な1:1の比率で動作される.
Cas9とgRNAが独立して、異なるコンストラクト上で発現するデュアルベクターは、マルチプレックスターゲティングでの複数gRNA発現を計画している際に最適です。これらのアプリケーションでは、Cas9は最初に細胞株で安定発現される必要があり、その後細胞に様々なgRNAベクターをトランスフェクトすることで、細胞プールを作ることができます.
CRISPR遺伝子編集用のベクター選択には、アプリケーションと細胞種両方を考慮する必要があります.
トランスフェクションが容易な細胞株の多くでは、非ウイルスベクターが良く機能できます。レンチウイルストランスフェクションは通常、一過性発現効率の低い細胞、例えばプライマリー細胞培養やトランスフェクションが困難な細胞株などで必要となります.
AAVベクターは免疫原性が低く、in vivoへの遺伝子導入に適しています。AAVベクターの積荷限界は他のベクターよりも一般的に小さく(<5 kb)、これらのベクターにSpCas9遺伝子をパッケージングすることは難しい場合があります。黄色ブドウ球菌Cas9オルソログ(SaCas9)はSpCas9よりも小さく、またAAVベクタ―に適したCas9バリアントです.
SpCas9ニッカーゼベクターは、オフターゲット編集により感受性のある実験での使用に利点があります。しかし、覚えておくべき重要な点として、順鎖と逆鎖の両方をターゲットするため、二つのgRNAをデザインする必要があるということです。これらのgRNAは、PAM部位がお互いに遠くになるよう、方向を合わせる必要があります。gRNA配列はそれらの間に、最大100 bpのウィンドウで埋め合わせる必要があります.
しっかりとしたSAM転写活性化に、gRNAは転写開始部位(TSS)の最初の200 bp上流をターゲットする必要があります。転写活性化の程度を低くするには、TSSの上流のより離れた距離に対するSAM複合体をターゲットするgRNAをデザインします.
転写を抑制するにはTSSに対して+50となるSAMをターゲットするgRNAをデザインすることで効果的にTSSをブロックしますGenScriptはゲノムワイドなSAM gRNAデータベースを持っています。このデータベースは、ヒトゲノムの各翻訳領域を活性化するためにデザインされた、6つのSAM gRNAを含んでいます。 他の種には、当社の科学者が無料のSAM gRNAデザイン支援を提供しています。
